Intro_FindIT2:mmAnno module
从这篇文章开始,我会做一个系列来介绍FindIT2包中每个模块的功能和应用场景。这篇文章介绍的是FindIT2的第一个也是最基本的模块:mmAnno module。mmAnno是multi-peak-multi-gene annotation的缩写,这也暗示了这个模块的主要功能:peak注释,或者说更严谨一点:建立peak与gene之间的link关系。
mmAnno模块现在有三个函数:mm_nearestGene,mm_geneScan以及mm_geneBound。这三个函数的设计初衷和应用场景各不相同,下面我会用测试数据来详细说明这三个函数的功能,同时还引入一些有用的辅助函数。
Pre-preparation
这篇文章的测试数据来自于FindIT2包,所以如果想要跟着一起练习的话,请下载并安装FindIT2包。同时由于我的测试数据是拟南芥,所以还需要下载拟南芥的Txdb包。
如果你的物种在Bioconductor上没有Txdb包,可以基于你自己物种的GTF或者GFF,通过
makeTxDbFromGFF来制作Txdb。详细内容请见:Making and Utilizing TxDb Objects
在后面的讲解中,我还会与Y叔的ChIPseeker包进行比较,所以这里还需要加载ChIPseeker包。同时由于我后面还需要有一些数据处理操作,所以还需要加载下dplyr包。
library(FindIT2)
library(TxDb.Athaliana.BioMart.plantsmart28)
library(ChIPseeker)
library(dplyr)
Txdb <- TxDb.Athaliana.BioMart.plantsmart28在加载完了包之后,我们还需要导入测试的peak文件,其来自于之前文章中的一个ChIP-seq数据1中第5条染色体的call peak结果。测试的peak文件地址在
ChIP_peak_path <- system.file("extdata", "ChIP.bed.gz", package = "FindIT2")
# 这是我电脑上的地址,你那里会是另外一个地址
ChIP_peak_path
## [1] "E:/R/R-4.1.0/library/FindIT2/extdata/ChIP.bed.gz"我们可以看下里面的内容
head(read.table(ChIP_peak_path))
## V1 V2 V3 V4 V5 V6
## 1 Chr5 6235 6508 peak_14125 27 .
## 2 Chr5 7626 8237 peak_14126 51 .
## 3 Chr5 9729 10211 peak_14127 32 .
## 4 Chr5 12692 12867 peak_14128 22 .
## 5 Chr5 13167 14770 peak_14129 519 .
## 6 Chr5 14854 16318 peak_14130 431 .这个文件是一个标准的6列bed文件,第1列是location,第2,3列是起始和终止,第4列是名字,第5列是score,第6列是strand。第4列尤为重要,FindIT2的几乎所有函数都要求你的peak文件有一列name列作为你peak的唯一标识,如果你的peak文件没有name列,也可以自己手动添加,这一点会在后面解释。
知道了peak地址,我们就需要将peak文件导入并保存成对象。在FindIT2包的所有函数中,凡是跟peak location相关的对象,一律都是GRanges对象,同时这也是Bioconductor自己官方的Genomic coordinates class。所以我们这里首先需要读取peak文件并将其信息转换成GRanges对象,FindIT2提供了一个loadPeakFile函数来帮助用户读取peak文件。
这里的loadPeakFile函数其实就是rtracklayer包中import函数的简单封装,只不过loadPeakFile将metadata columns中的第一列,即前面的第4列,name列,的名字改成了feature_id。feature_id这个列名以及对应的列信息是非常重要的!!!!,如果你没有这一列或者列名不是feature_id,后面的很多函数就会报错,所以请再次确保你有这一列及列名。
关于Granges对象或者metadata column的相关信息,请参见GenomicRanges包的手册。
# rtracklayer::import函数的结果
rtracklayer::import(ChIP_peak_path)
## GRanges object with 4288 ranges and 2 metadata columns:
## seqnames ranges strand | name score
## <Rle> <IRanges> <Rle> | <character> <numeric>
## [1] Chr5 6236-6508 * | peak_14125 27
## [2] Chr5 7627-8237 * | peak_14126 51
## [3] Chr5 9730-10211 * | peak_14127 32
## [4] Chr5 12693-12867 * | peak_14128 22
## [5] Chr5 13168-14770 * | peak_14129 519
## ... ... ... ... . ... ...
## [4284] Chr5 26937822-26938526 * | peak_18408 445
## [4285] Chr5 26939152-26939267 * | peak_18409 21
## [4286] Chr5 26949581-26950335 * | peak_18410 263
## [4287] Chr5 26952230-26952558 * | peak_18411 30
## [4288] Chr5 26968877-26969091 * | peak_18412 26
## -------
## seqinfo: 1 sequence from an unspecified genome; no seqlengths# 我们用loadPeakFile函数导入peak文件,并保存成一个对象
# 原来的|旁边的第一列的列名从name变成了feature_id
ChIP_peak_GR <- loadPeakFile(ChIP_peak_path)
ChIP_peak_GR
## GRanges object with 4288 ranges and 2 metadata columns:
## seqnames ranges strand | feature_id score
## <Rle> <IRanges> <Rle> | <character> <numeric>
## [1] Chr5 6236-6508 * | peak_14125 27
## [2] Chr5 7627-8237 * | peak_14126 51
## [3] Chr5 9730-10211 * | peak_14127 32
## [4] Chr5 12693-12867 * | peak_14128 22
## [5] Chr5 13168-14770 * | peak_14129 519
## ... ... ... ... . ... ...
## [4284] Chr5 26937822-26938526 * | peak_18408 445
## [4285] Chr5 26939152-26939267 * | peak_18409 21
## [4286] Chr5 26949581-26950335 * | peak_18410 263
## [4287] Chr5 26952230-26952558 * | peak_18411 30
## [4288] Chr5 26968877-26969091 * | peak_18412 26
## -------
## seqinfo: 1 sequence from an unspecified genome; no seqlengths如果你认为你的name列不存在,你也可以手动自己添加。
# 这里我直接手动添加了一列feature_id
# 当然你也可以自己手动更改name列的列名,具体操作详见GenomicRanges的手册
peakGR_test <- rtracklayer::import(ChIP_peak_path)
peakGR_test$feature_id <- paste0("peak", seq_len(length(peakGR_test)))
peakGR_test
## GRanges object with 4288 ranges and 3 metadata columns:
## seqnames ranges strand | name score feature_id
## <Rle> <IRanges> <Rle> | <character> <numeric> <character>
## [1] Chr5 6236-6508 * | peak_14125 27 peak1
## [2] Chr5 7627-8237 * | peak_14126 51 peak2
## [3] Chr5 9730-10211 * | peak_14127 32 peak3
## [4] Chr5 12693-12867 * | peak_14128 22 peak4
## [5] Chr5 13168-14770 * | peak_14129 519 peak5
## ... ... ... ... . ... ... ...
## [4284] Chr5 26937822-26938526 * | peak_18408 445 peak4284
## [4285] Chr5 26939152-26939267 * | peak_18409 21 peak4285
## [4286] Chr5 26949581-26950335 * | peak_18410 263 peak4286
## [4287] Chr5 26952230-26952558 * | peak_18411 30 peak4287
## [4288] Chr5 26968877-26969091 * | peak_18412 26 peak4288
## -------
## seqinfo: 1 sequence from an unspecified genome; no seqlengths这里还有一个小问题就是我们Txdb中的染色体信息是以1,2,3,4,5,Mt,Pt这样的信息存在的,而我们的ChIP_peak_GR中的染色体信息则是Chr5。所以我们需要修改其中一个对象的染色体信息,这里我选择修改Txdb的。
seqlevels(Txdb)
## [1] "1" "2" "3" "4" "5" "Mt" "Pt"
seqlevels(ChIP_peak_GR)
## [1] "Chr5"
seqlevels(Txdb) <- paste0("Chr", c(1:5, "M", "C"))
seqlevels(Txdb)
## [1] "Chr1" "Chr2" "Chr3" "Chr4" "Chr5" "ChrM" "ChrC"mm_nearestGene
mm_nearestGene这个函数从名字上就可以看出其执行的是最近基因注释原则,这也是现在peak注释中最普遍的一个原则。最近基因注释原则顾名思义就是在执行peak注释的时候,挑选距离peak最近的那个基因作为peak的target基因,从而为这个peak添加相关的信息。ChIPseeker、ChIPpeakAnno等包的基本注释也都是应用这个原则,只不过在注释的细节、逻辑及参数调整上会略有不同,从而使得软件之间最终的结果会有细微差别。
mm_nearestGene是GenomicRanges包中distanceToNearest函数的进一步封装,其基本逻辑就是提取Txdb数据包中的基因TSS位置,然后为你输入的每个peak寻找距离最近的那个基因TSS,从而将peak与对应的基因关联起来。
mmAnno_nearestgene <- mm_nearestGene(peak_GR = ChIP_peak_GR,
Txdb = Txdb)
## >> checking seqlevels match... 2021-08-11 20:27:28
## >> your peak_GR seqlevel:Chr5...
## >> your Txdb seqlevel:Chr1 Chr2 Chr3 Chr4 Chr5 ChrM ChrC...
## Good, your Chrs in peak_GR is all in Txdb
## ------------
## annotating Peak using nearest gene mode begins
## >> preparing gene features information... 2021-08-11 20:27:28
## >> finding nearest gene and calculating distance... 2021-08-11 20:27:29
## >> dealing with gene strand ... 2021-08-11 20:27:29
## >> merging all info together ... 2021-08-11 20:27:29
## >> done 2021-08-11 20:27:29可以看到,mm_nearestGene的输出结果仍旧是一个GRanges对象,只不过会在原有的基础上再加额外的几列信息,分别是距离最近的那个基因,以及距离基因TSS的位置。负号代表这个peak在基因的上游,正号代表在基因的下游,0代表这个peak跟基因TSS有overlap。除了直观的每个peak的注释结果,FindIT2还提供了一些辅助函数来帮助用户更好地查看结果。
有时候我们还想看下peak在基因组上距离的整体分布,从而来判断你TF的属性:是promoter-type还是enhancer type的。promoter-type的TF其会偏向于定位在基因TSS附近而enhancer type的则会偏向于定位在基因间区,即远离基因TSS。针对这个需求,FindIT2还提供了一个函数plot_annoDistance,其可以画出distanceToTSS的概述图。从下图可以看到,这TF主要有两个峰,主峰是在0bp那里,意味着大部分的peak都是在TSS附近的,所以这个TF应该是一个promoter-type的TF。
plot_annoDistance(mmAnno = mmAnno_nearestgene)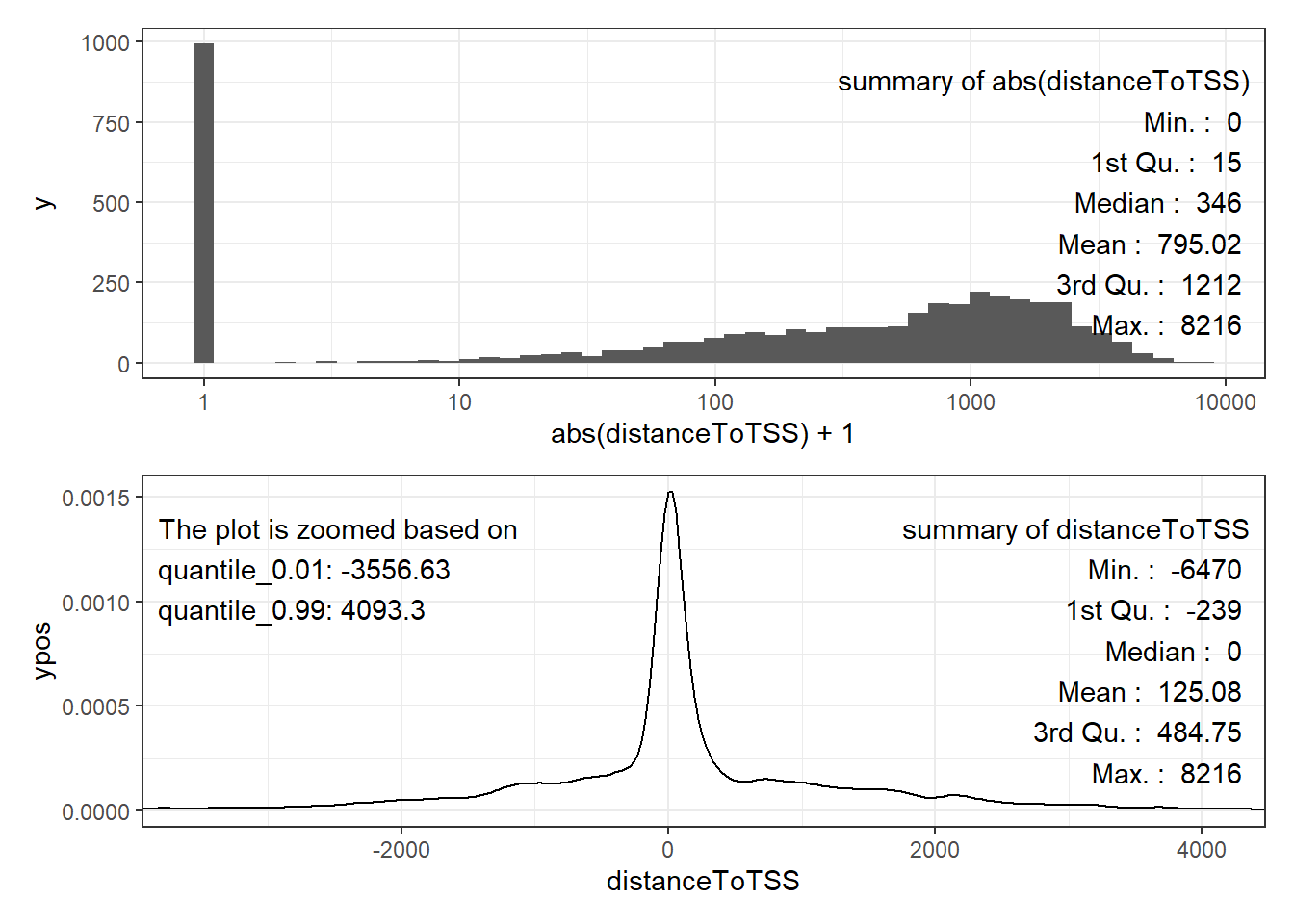
除了对peak距离的整体概述,很多人还会对基因临近的peak数目感兴趣,如果一个基因附近的peak数目越多,说明这个基因周围更可能具有复杂调控机制。针对这个目的,FindIT2提供了getAssocPairNumber函数,其可以告诉用户每个基因对应的peak数目。
getAssocPairNumber(mmAnno = mmAnno_nearestgene)
## # A tibble: 2,757 x 2
## gene_id peakNumber
## <chr> <int>
## 1 AT5G01015 1
## 2 AT5G01020 1
## 3 AT5G01030 2
## 4 AT5G01040 2
## 5 AT5G01050 2
## 6 AT5G01070 1
## 7 AT5G01090 1
## 8 AT5G01100 1
## 9 AT5G01170 1
## 10 AT5G01175 3
## # ... with 2,747 more rows
# 如果想要排序的结果,可以用dplyr包的arrange函数
getAssocPairNumber(mmAnno = mmAnno_nearestgene) %>%
arrange(desc(peakNumber))
## # A tibble: 2,757 x 2
## gene_id peakNumber
## <chr> <int>
## 1 AT5G05140 8
## 2 AT5G17810 8
## 3 AT5G53290 8
## 4 AT5G13170 7
## 5 AT5G25810 7
## 6 AT5G53470 7
## 7 AT5G67190 7
## 8 AT5G04310 6
## 9 AT5G24030 6
## 10 AT5G25220 6
## # ... with 2,747 more rows在得到了每个基因的详细peak数目之后,我们可能还得整体的统计数字感兴趣,这时候你只需要将output_output_summary参数改成TRUE即可。从下面结果可以看到,大部分基因的临近peak都是1个,只有几个基因其临近peak数目是大于8的。
getAssocPairNumber(mmAnno = mmAnno_nearestgene,
output_summary = TRUE)
## # A tibble: 8 x 2
## N gene_freq
## <fct> <int>
## 1 >=8 3
## 2 1 1793
## 3 2 606
## 4 3 229
## 5 4 75
## 6 5 38
## 7 6 9
## 8 7 4除了文字的描述之外,你可能还想要有图片的概述。FindIT2提供了plot_peakGeneAlias_summary函数来帮你直观地展现这个结果。
plot_peakGeneAlias_summary(mmAnno = mmAnno_nearestgene)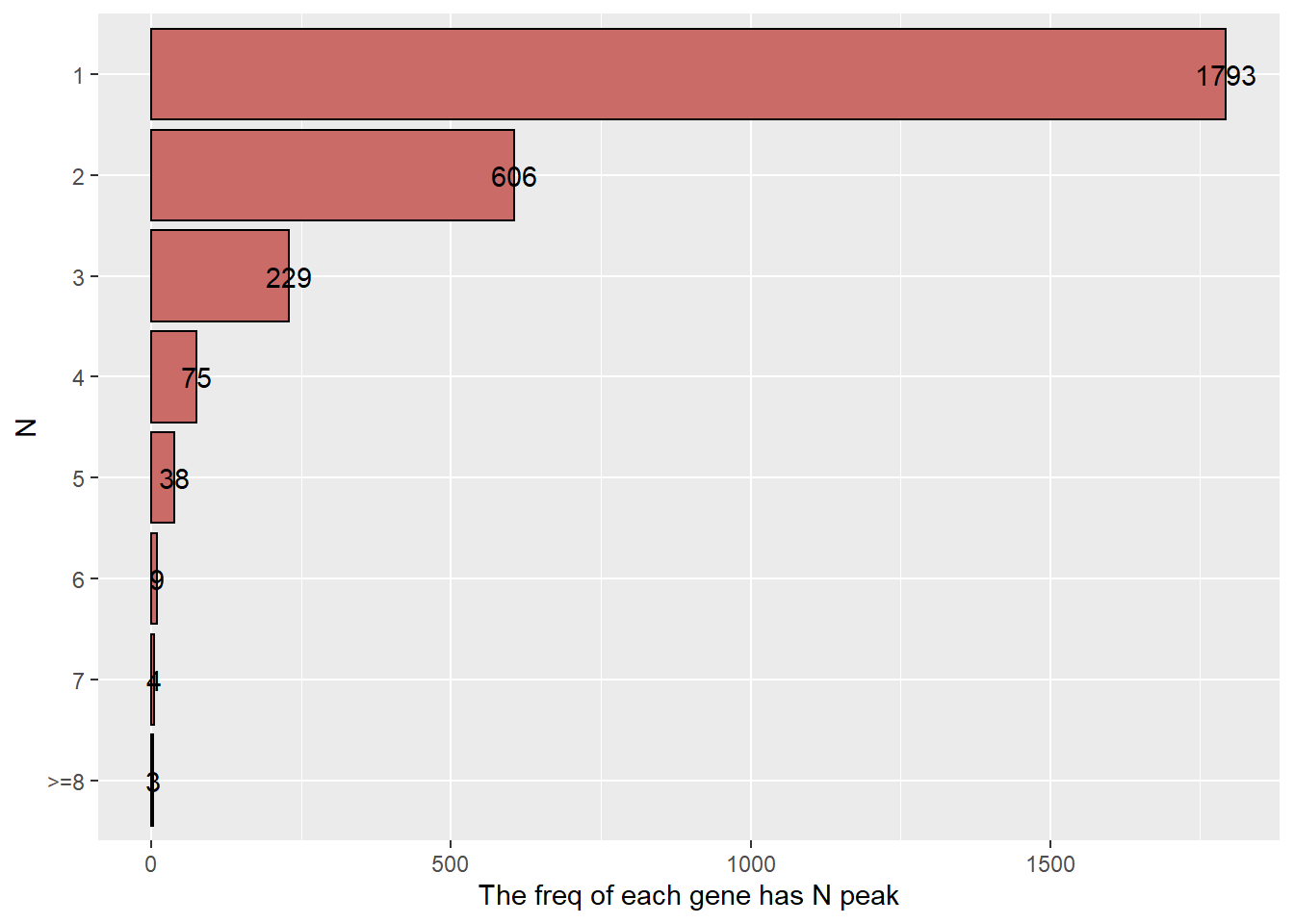
我在前面提到过,现在大部分peak注释软件的基本注释模式是最近基因注释原则,只不过由于一些参数细节上的微调,使得最终的结果有些许不同。我这里拿ChIPseeker的注释结果来做比较
ChIPseeker_anno <- annotatePeak(peak = ChIP_peak_GR,
TxDb = Txdb,
level = "gene")
## >> preparing features information... 2021-08-11 20:27:30
## >> identifying nearest features... 2021-08-11 20:27:31
## >> calculating distance from peak to TSS... 2021-08-11 20:27:31
## >> assigning genomic annotation... 2021-08-11 20:27:31
## >> assigning chromosome lengths 2021-08-11 20:27:35
## >> done... 2021-08-11 20:27:35
ChIPseeker_anno <- ChIPseeker_anno@anno最直观的比较方法就是看peak-gene pair以及对应的距离结果是否相同
annoPair_ChIPseeker <- paste(ChIPseeker_anno$feature_id,
ChIPseeker_anno$geneId,
ChIPseeker_anno$distanceToTSS,
sep = "/")
head(annoPair_ChIPseeker)
## [1] "peak_14125/AT5G01015/-345" "peak_14126/AT5G01020/207"
## [3] "peak_14127/AT5G01030/0" "peak_14128/AT5G01030/2824"
## [5] "peak_14129/AT5G01040/1403" "peak_14130/AT5G01040/0"
annoPair_mm <- paste(mmAnno_nearestgene$feature_id,
mmAnno_nearestgene$gene_id,
mmAnno_nearestgene$distanceToTSS,
sep = "/")
head(annoPair_mm)
## [1] "peak_14125/AT5G01015/-344" "peak_14126/AT5G01020/206"
## [3] "peak_14127/AT5G01030/0" "peak_14128/AT5G01030/2823"
## [5] "peak_14129/AT5G01040/1402" "peak_14130/AT5G01040/0"从前几个结果来看,mm_nearestGene的结果与ChIPseeker的结果是很类似的,只不过在距离上要比ChIPseeker少1bp而已,这个是跟Y叔对于距离的定义有关。所以我们稍微修改下用来对照的结果,即只看peak-gene pair。
annoPair_ChIPseeker <- paste(ChIPseeker_anno$feature_id,
ChIPseeker_anno$geneId,
sep = "/")
annoPair_mm <- paste(mmAnno_nearestgene$feature_id,
mmAnno_nearestgene$gene_id,
sep = "/")
mean(annoPair_mm %in% annoPair_ChIPseeker)
## [1] 0.9874067从上面的结果可以看出,mm_nearestGene大约有55个结果(4288*0.013)是和ChIPseeker不对应的,我们可以具体地来看看是哪些结果。可以看到,对于某些peak,ChIPseeker和mm_nearestGene给出的target gene是不一致的。
annoPair_mm[!annoPair_mm %in% annoPair_ChIPseeker]
## [1] "peak_14179/AT5G01712" "peak_14213/AT5G01960" "peak_14228/AT5G02240"
## [4] "peak_14276/AT5G02820" "peak_14294/AT5G03080" "peak_14780/AT5G09445"
## [7] "peak_14781/AT5G09463" "peak_14782/AT5G09463" "peak_14783/AT5G09463"
## [10] "peak_14784/AT5G09460" "peak_14853/AT5G10460" "peak_14879/AT5G10830"
## [13] "peak_15177/AT5G14560" "peak_15510/AT5G19230" "peak_15546/AT5G19730"
## [16] "peak_15633/AT5G20800" "peak_15663/AT5G21430" "peak_15723/AT5G22545"
## [19] "peak_15925/AT5G25520" "peak_15935/AT5G25754" "peak_16038/AT5G27410"
## [22] "peak_16168/AT5G36925" "peak_16216/AT5G38160" "peak_16287/AT5G39581"
## [25] "peak_16359/AT5G40855" "peak_16444/AT5G42053" "peak_16681/AT5G45430"
## [28] "peak_16701/AT5G45750" "peak_16806/AT5G47230" "peak_17010/AT5G50361"
## [31] "peak_17039/AT5G51120" "peak_17245/AT5G53590" "peak_17347/AT5G54970"
## [34] "peak_17360/AT5G55055" "peak_17386/AT5G55630" "peak_17533/AT5G57391"
## [37] "peak_17618/AT5G58340" "peak_17630/AT5G58450" "peak_17646/AT5G58660"
## [40] "peak_17714/AT5G59732" "peak_17717/AT5G59750" "peak_17731/AT5G59920"
## [43] "peak_17737/AT5G59970" "peak_17822/AT5G60963" "peak_17835/AT5G61230"
## [46] "peak_18066/AT5G64343" "peak_18079/AT5G64505" "peak_18084/AT5G64550"
## [49] "peak_18085/AT5G64550" "peak_18135/AT5G65120" "peak_18168/AT5G65360"
## [52] "peak_18175/AT5G65450" "peak_18330/AT5G66930" "peak_18405/AT5G67500"
annoPair_ChIPseeker[!annoPair_ChIPseeker %in% annoPair_mm]
## [1] "peak_14179/AT5G01710" "peak_14213/AT5G01950" "peak_14228/AT5G02230"
## [4] "peak_14276/AT5G02811" "peak_14294/AT5G03070" "peak_14780/AT5G09443"
## [7] "peak_14781/AT5G09460" "peak_14782/AT5G09460" "peak_14783/AT5G09460"
## [10] "peak_14784/AT5G09463" "peak_14853/AT5G10455" "peak_14879/AT5G10820"
## [13] "peak_15177/AT5G14550" "peak_15510/AT5G19220" "peak_15546/AT5G19729"
## [16] "peak_15633/AT5G20790" "peak_15663/AT5G21378" "peak_15723/AT5G22540"
## [19] "peak_15925/AT5G25510" "peak_15935/AT5G25752" "peak_16038/AT5G27400"
## [22] "peak_16168/AT5G36920" "peak_16216/AT5G38155" "peak_16287/AT5G39580"
## [25] "peak_16359/AT5G40850" "peak_16444/AT5G42050" "peak_16681/AT5G45428"
## [28] "peak_16701/AT5G45745" "peak_16806/AT5G47229" "peak_17010/AT5G50360"
## [31] "peak_17039/AT5G51110" "peak_17245/AT5G53588" "peak_17347/AT5G54960"
## [34] "peak_17360/AT5G55045" "peak_17386/AT5G55620" "peak_17533/AT5G57390"
## [37] "peak_17618/AT5G58330" "peak_17630/AT5G58440" "peak_17646/AT5G58650"
## [40] "peak_17714/AT5G59730" "peak_17717/AT5G59740" "peak_17731/AT5G59910"
## [43] "peak_17737/AT5G59960" "peak_17822/AT5G60960" "peak_17835/AT5G61228"
## [46] "peak_18066/AT5G64340" "peak_18079/AT5G64501" "peak_18084/AT5G64552"
## [49] "peak_18085/AT5G64552" "peak_18135/AT5G65110" "peak_18168/AT5G65350"
## [52] "peak_18175/AT5G65445" "peak_18330/AT5G66920" "peak_18405/AT5G67488"我们可以挑一个peak来详细地看看它周边的基因情况。以peak_14213为例。mm_nearestGene给出的是AT5G01960,而ChIPseeker给出的是AT5G01950,如果我们去IGV里面查看的话,可以看到这个peak周围有非常致密的基因区域。针对这种情况,似乎用AT5G01950或者AT5G01960去注释peak_14213这个peak都是合理的。
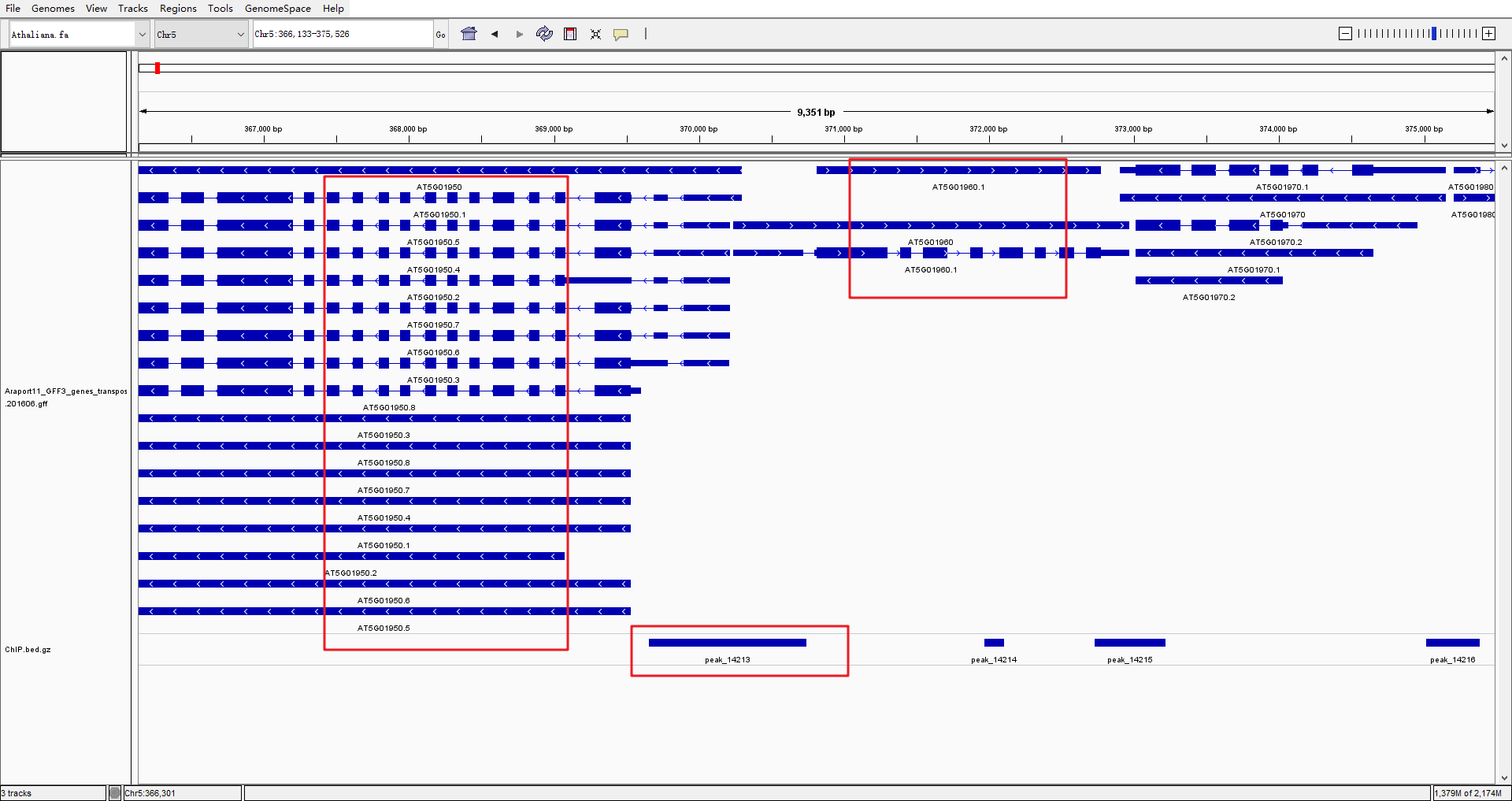
事实上,这种情况在小基因组中尤为的常见。已有的生物学常识告诉我们大部分的物种基因数目都是稳定在3w左右,但基因组大小却可以有几个数量级的区别,基因组越小,那么基因间区就会越小,对应的致密基因区域就会越多,这时候peak注释就会存在一些问题,因为可能peak分配给哪个基因都是合理的。由此,这也引出了下一个函数:mm_geneScan。
mm_geneScan
上面的mm_nearestGene所产生的结果其实是一对一的,即一个peak只会被分配给一个基因,换句话说,如果一个peak被一个基因注释了,那么它就不会再被另一个基因所注释了。但这个注释逻辑在有些时候是存在问题的,最直观地就是上面peak_14213的例子。这个peak其实可以既可以分配给AT5G01950又可以分配给AT5G01960,甚至临近的其他基因,但由于分配逻辑的缘故,使得该peak只会被注释一次。基于这个原因,我写了另一个函数mm_geneScan。
其基本的逻辑就是提取Txdb数据包中的基因TSS位置,然后根据你设置的upstream和downstream参数来形成一个扫描区域,凡是落在这个扫描区域内的peak都会与对应的基因形成peak-gene pair。如果有些peak没有落在任何一个扫描区域内,就再次执行最近基因注释原则,为这些peak寻找最近的那个基因。通过这个注释逻辑,我们最终所产生的就是多对多的注释结果,即一个peak会被分配给多个基因。
# 我这里设置上下游各3000bp作为扫描区域,你也可以根据自己的偏好设置
# 区域越大,对应的peak-gene pair就越多
# 区域参数如果接近0的话,最终的结果就会跟mm_nearestGene类似了
mmAnno_geneScan <- mm_geneScan(peak_GR = ChIP_peak_GR,
Txdb = Txdb,
upstream = 3000,
downstream = 3000)
## >> checking seqlevels match... 2021-08-11 20:27:35
## >> your peak_GR seqlevel:Chr5...
## >> your Txdb seqlevel:Chr1 Chr2 Chr3 Chr4 Chr5 ChrM ChrC...
## Good, your Chrs in peak_GR is all in Txdb
## ------------
## annotatePeak using geneScan mode begins
## >> preparing gene features information and scan region... 2021-08-11 20:27:35
## >> some scan range may cross Chr bound, trimming... 2021-08-11 20:27:36
## >> finding overlap peak in gene scan region... 2021-08-11 20:27:36
## >> dealing with left peak not your gene scan region... 2021-08-11 20:27:36
## >> merging two set peaks... 2021-08-11 20:27:36
## >> calculating distance and dealing with gene strand... 2021-08-11 20:27:36
## >> merging all info together ... 2021-08-11 20:27:36
## >> done 2021-08-11 20:27:36
mmAnno_geneScan
## GRanges object with 8419 ranges and 4 metadata columns:
## seqnames ranges strand | feature_id score gene_id
## <Rle> <IRanges> <Rle> | <character> <numeric> <character>
## [1] Chr5 6236-6508 * | peak_14125 27 AT5G01010
## [2] Chr5 7627-8237 * | peak_14126 51 AT5G01010
## [3] Chr5 6236-6508 * | peak_14125 27 AT5G01015
## [4] Chr5 7627-8237 * | peak_14126 51 AT5G01015
## [5] Chr5 6236-6508 * | peak_14125 27 AT5G01020
## ... ... ... ... . ... ... ...
## [8415] Chr5 26284851-26285670 * | peak_18207 67 AT5G65700
## [8416] Chr5 26318831-26319147 * | peak_18210 89 AT5G65790
## [8417] Chr5 26487154-26487324 * | peak_18260 20 AT5G66310
## [8418] Chr5 26628525-26628676 * | peak_18307 24 AT5G66690
## [8419] Chr5 26812830-26813041 * | peak_18357 30 AT5G67190
## distanceToTSS
## <numeric>
## [1] -1174
## [2] -2565
## [3] -344
## [4] -1735
## [5] 1935
## ... ...
## [8415] 3139
## [8416] -3800
## [8417] 4057
## [8418] 3444
## [8419] -3140
## -------
## seqinfo: 1 sequence from an unspecified genome; no seqlengths可以看到,mmAnno_geneScan的输出结果仍旧是GRanges格式,这就意味前面mm_nearestGene相关的辅助函数都可以应用在这里,除了一个函数:plot_annoDistance。因为我觉得多对多的情况下画距离概述图是没有任何意义的。
plot_annoDistance(mmAnno = mmAnno_geneScan)
## Error: sorry, it only accept mmAnno from mm_nearestGene我们可以列出每个基因对应的peak数目。
getAssocPairNumber(mmAnno = mmAnno_geneScan)
## # A tibble: 4,317 x 2
## gene_id peakNumber
## <chr> <int>
## 1 AT5G01010 2
## 2 AT5G01015 2
## 3 AT5G01020 3
## 4 AT5G01030 3
## 5 AT5G01040 2
## 6 AT5G01050 2
## 7 AT5G01060 2
## 8 AT5G01070 1
## 9 AT5G01075 1
## 10 AT5G01080 2
## # ... with 4,307 more rows同时,getAssocPairNumber还可以切换成计算每个peak对应的基因数目
getAssocPairNumber(mmAnno = mmAnno_geneScan,
output_type = "feature_id")
## # A tibble: 4,288 x 2
## feature_id geneNumber
## <chr> <int>
## 1 peak_14125 3
## 2 peak_14126 4
## 3 peak_14127 2
## 4 peak_14128 1
## 5 peak_14129 1
## 6 peak_14130 1
## 7 peak_14131 2
## 8 peak_14132 2
## 9 peak_14133 3
## 10 peak_14134 2
## # ... with 4,278 more rows
# 如果你输入mmAnno_nearestGene
# 就会发现peak对应的基因数目都是1,因为其执行的是一对一的注释逻辑
getAssocPairNumber(mmAnno = mmAnno_nearestgene,
output_type = "feature_id")
## # A tibble: 4,288 x 2
## feature_id geneNumber
## <chr> <int>
## 1 peak_14125 1
## 2 peak_14126 1
## 3 peak_14127 1
## 4 peak_14128 1
## 5 peak_14129 1
## 6 peak_14130 1
## 7 peak_14131 1
## 8 peak_14132 1
## 9 peak_14133 1
## 10 peak_14134 1
## # ... with 4,278 more rows我们还可以得到整体的统计数字以及图像。
getAssocPairNumber(mmAnno = mmAnno_geneScan,
output_type = "gene_id",
output_summary = TRUE)
## # A tibble: 8 x 2
## N gene_freq
## <fct> <int>
## 1 >=8 4
## 2 1 2051
## 3 2 1168
## 4 3 614
## 5 4 310
## 6 5 116
## 7 6 40
## 8 7 14
getAssocPairNumber(mmAnno = mmAnno_geneScan,
output_type = "feature_id",
output_summary = TRUE)
## # A tibble: 7 x 2
## N feature_freq
## <fct> <int>
## 1 1 1599
## 2 2 1606
## 3 3 788
## 4 4 244
## 5 5 42
## 6 6 5
## 7 7 4plot_peakGeneAlias_summary(mmAnno = mmAnno_geneScan,
output_type = "gene_id")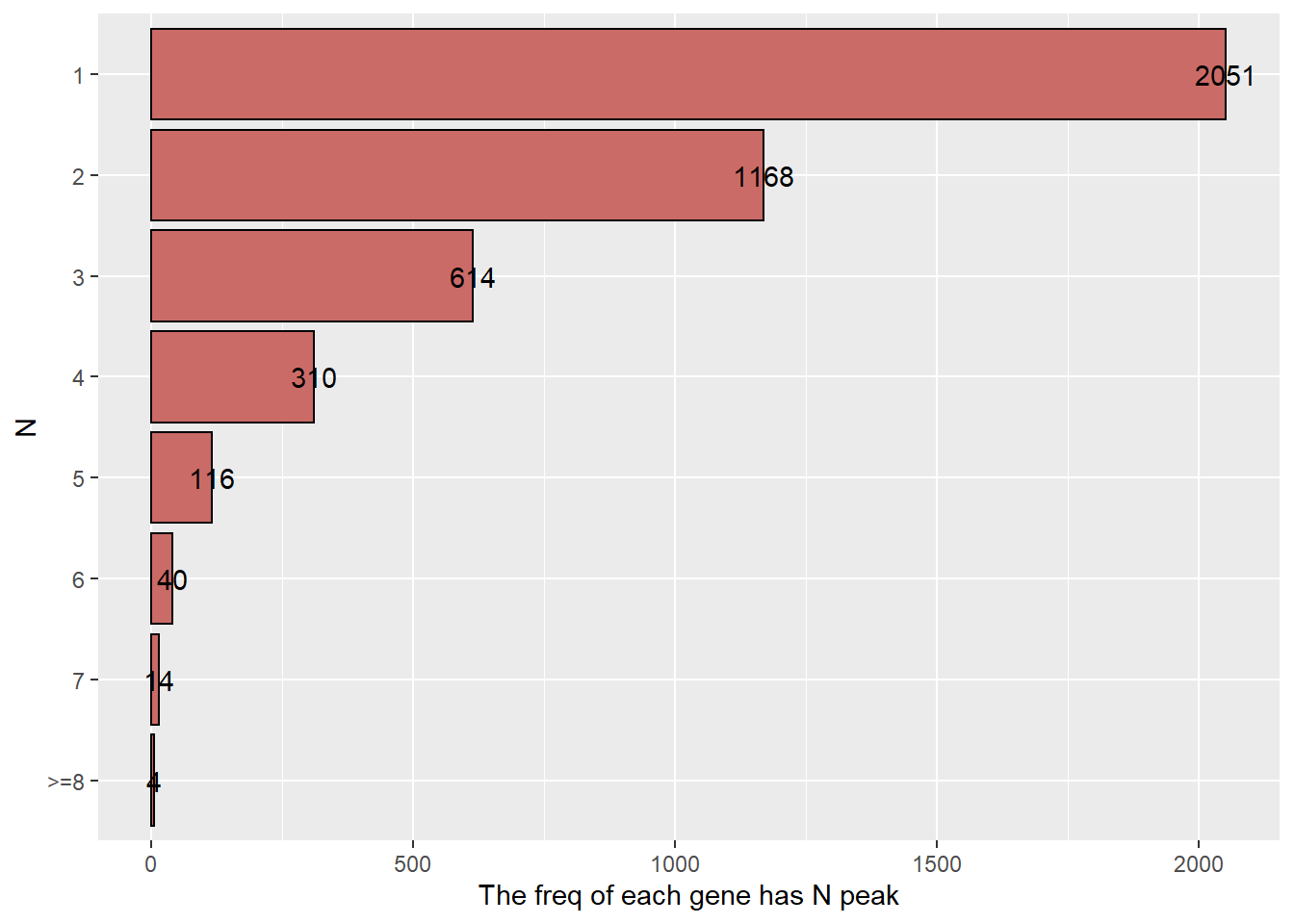
plot_peakGeneAlias_summary(mmAnno = mmAnno_geneScan,
output_type = "feature_id")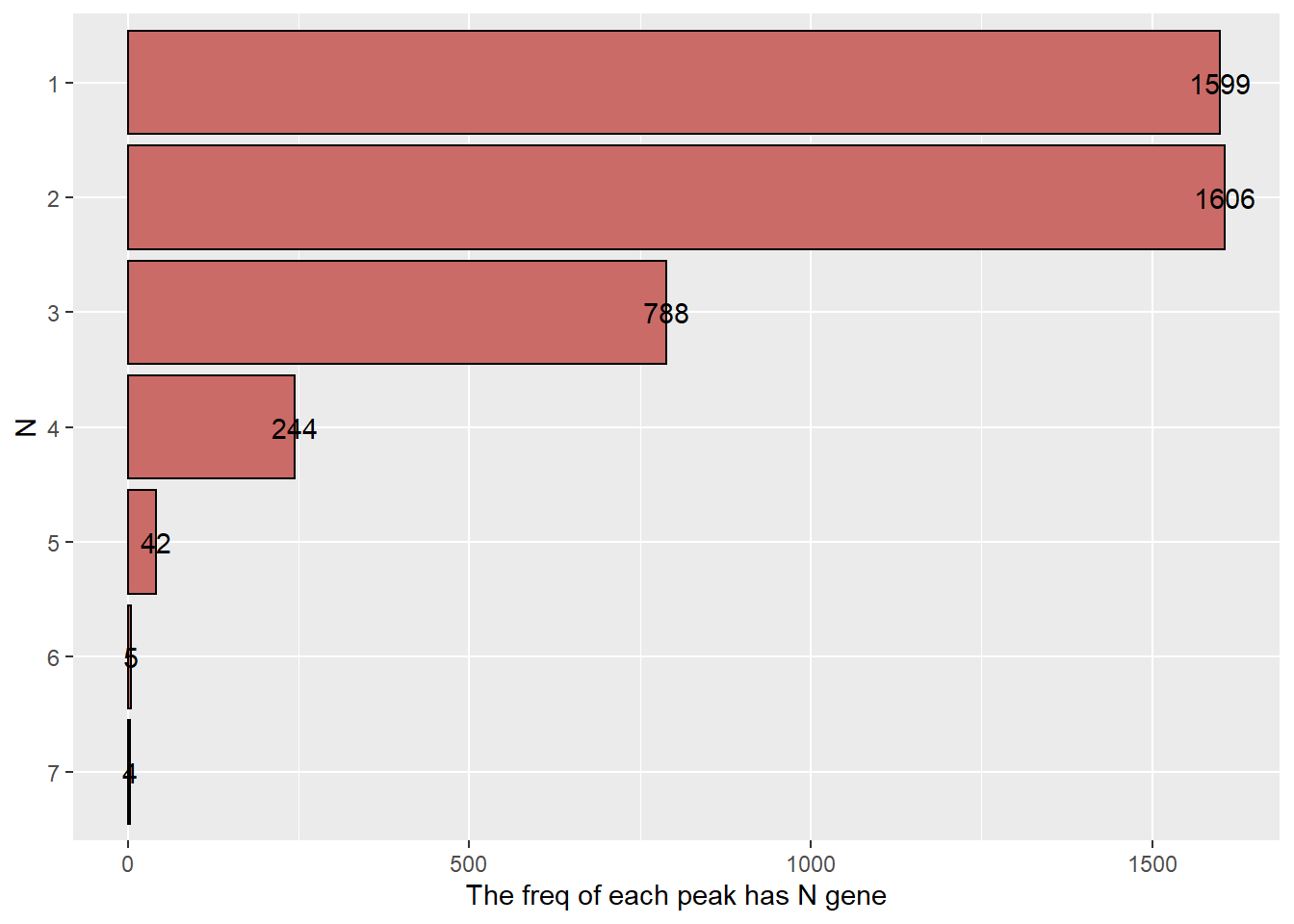
前面我提到了mm_nearestGene和ChIPseeker的注释结果有一些不一致,这里我们来看看mm_geneScan的结果是否能够解决这个矛盾。
annoPair_mmScan <- paste(mmAnno_geneScan$feature_id,
mmAnno_geneScan$gene_id,
sep = "/")
mean(annoPair_mm %in% annoPair_mmScan)
## [1] 1
mean(annoPair_ChIPseeker %in% annoPair_mmScan)
## [1] 1可以看到,不管是mm_nearestGene或是ChIPseeker的结果,都是包含在mm_geneScan的结果中的。然后我们再来看看peak_14213相关的注释。通过下面的结果,我们可以看到mm_geneScan把两个基因注释结果都包含了。
subset(mmAnno_geneScan, feature_id == "peak_14213")
## GRanges object with 2 ranges and 4 metadata columns:
## seqnames ranges strand | feature_id score gene_id
## <Rle> <IRanges> <Rle> | <character> <numeric> <character>
## [1] Chr5 369657-370744 * | peak_14213 90 AT5G01950
## [2] Chr5 369657-370744 * | peak_14213 90 AT5G01960
## distanceToTSS
## <numeric>
## [1] 0
## [2] 0
## -------
## seqinfo: 1 sequence from an unspecified genome; no seqlengths事实上,mm_geneScan是整个FindIT2包中最重要也是最基本的函数,后续的很多函数的输入都依赖于其输出结果,我在后面的文章中也会不断地提到这个函数以及解释其应用场景。
mm_geneBound
mm_geneBound的设计目的主要是为你输入的基因寻找关联的peak,应用场景主要在画peak的热图或者火山图中。在画上述的图像中,我们往往会挑选一些感兴趣的基因来展示其相关peak的表达量变化或者差异倍数变化。但传统的最近注释原则由于其一对一的特性,会使得很多基因没有关联的peak,从而就会在热图或者火山图中缺失。如果只有2,3个基因缺失,你可以人为地挑选相关的peak,但如果有数十个基因的话,肉眼在IGV上寻找相关peak是一件非常痛苦的事情。而如果用mm_geneScan的结果去输出对应peak的话,也会存在一些问题。如果你scan范围设置的小了,你感兴趣的基因不一定会有相关的peak,如果scan范围设置的大了,那么你感兴趣的基因就会有非常多的关联peak,那么热图的篇幅机会很大,火山图上也会有密密麻麻的注释点。
所以针对上面的问题,我写了mm_geneBound。这个函数的注释原则非常的简单,其会首先执行最近基因注释原则,然后为哪些没有关联peak的基因执行最近peak注释原则,即找到距离基因TSS最近的peak。这样就保证了你每个基因都会至少有一个关联的peak。
peakGene_pair <- mm_geneBound(peak_GR = ChIP_peak_GR,
Txdb = Txdb,
input_genes = c("AT5G01015", "AT5G67570"))
## >> checking seqlevels match... 2021-08-11 20:27:37
## >> your peak_GR seqlevel:Chr5...
## >> your Txdb seqlevel:Chr1 Chr2 Chr3 Chr4 Chr5 ChrM ChrC...
## Good, your Chrs in peak_GR is all in Txdb
## >> using mm_nearestGene to annotate Peak... 2021-08-11 20:27:37
## >> checking seqlevels match... 2021-08-11 20:27:37
## >> your peak_GR seqlevel:Chr5...
## >> your Txdb seqlevel:Chr1 Chr2 Chr3 Chr4 Chr5 ChrM ChrC...
## Good, your Chrs in peak_GR is all in Txdb
## ------------
## annotating Peak using nearest gene mode begins
## >> preparing gene features information... 2021-08-11 20:27:37
## >> finding nearest gene and calculating distance... 2021-08-11 20:27:37
## >> dealing with gene strand ... 2021-08-11 20:27:38
## >> merging all info together ... 2021-08-11 20:27:38
## >> done 2021-08-11 20:27:38
## all your input gene have been annotated by nearestGene mode
peakGene_pair
## # A tibble: 2 x 3
## feature_id gene_id distanceToTSS_abs
## <chr> <chr> <dbl>
## 1 peak_14125 AT5G01015 344
## 2 peak_18411 AT5G67570 0可以看到,其输出结果是一个三列的tibble格式,这也方便用户自己再进一步地添加相关的peak-gene pair。
Session info
## - Session info -------------------------------------------------------------------------------------------------------
## setting value
## version R version 4.1.0 (2021-05-18)
## os Windows 10 x64
## system x86_64, mingw32
## ui RTerm
## language (EN)
## collate Chinese (Simplified)_China.936
## ctype Chinese (Simplified)_China.936
## tz Asia/Taipei
## date 2021-08-11
##
## - Packages -----------------------------------------------------------------------------------------------------------
## package * version date lib source
## AnnotationDbi * 1.54.1 2021-06-08 [1] Bioconductor
## ape 5.5 2021-04-25 [1] CRAN (R 4.1.0)
## aplot 0.0.6 2020-09-03 [1] CRAN (R 4.1.0)
## assertthat 0.2.1 2019-03-21 [1] CRAN (R 4.1.0)
## Biobase * 2.52.0 2021-05-19 [1] Bioconductor
## BiocFileCache 2.0.0 2021-05-19 [1] Bioconductor
## BiocGenerics * 0.38.0 2021-05-19 [1] Bioconductor
## BiocIO 1.2.0 2021-05-19 [1] Bioconductor
## BiocManager 1.30.15 2021-05-11 [1] CRAN (R 4.1.0)
## BiocParallel 1.26.0 2021-05-19 [1] Bioconductor
## biomaRt 2.48.2 2021-07-01 [1] Bioconductor
## Biostrings 2.60.0 2021-05-20 [1] Bioconductor
## bit 4.0.4 2020-08-04 [1] CRAN (R 4.1.0)
## bit64 4.0.5 2020-08-30 [1] CRAN (R 4.1.0)
## bitops 1.0-7 2021-04-24 [1] CRAN (R 4.1.0)
## blob 1.2.2 2021-07-23 [1] CRAN (R 4.1.0)
## blogdown 1.4 2021-07-23 [1] CRAN (R 4.1.0)
## bookdown 0.22 2021-04-22 [1] CRAN (R 4.1.0)
## boot 1.3-28 2021-05-03 [1] CRAN (R 4.1.0)
## bslib 0.2.5.1 2021-05-18 [1] CRAN (R 4.1.0)
## cachem 1.0.5 2021-05-15 [1] CRAN (R 4.1.0)
## caTools 1.18.2 2021-03-28 [1] CRAN (R 4.1.0)
## ChIPseeker * 1.28.3 2021-05-21 [1] Bioconductor
## cli 3.0.1 2021-07-17 [1] CRAN (R 4.1.0)
## codetools 0.2-18 2020-11-04 [1] CRAN (R 4.1.0)
## colorspace 2.0-1 2021-05-04 [1] CRAN (R 4.1.0)
## cowplot 1.1.1 2020-12-30 [1] CRAN (R 4.1.0)
## crayon 1.4.1 2021-02-08 [1] CRAN (R 4.1.0)
## curl 4.3.1 2021-04-30 [1] CRAN (R 4.1.0)
## data.table 1.14.0 2021-02-21 [1] CRAN (R 4.1.0)
## DBI 1.1.1 2021-01-15 [1] CRAN (R 4.1.0)
## dbplyr 2.1.1 2021-04-06 [1] CRAN (R 4.1.0)
## DelayedArray 0.18.0 2021-05-19 [1] Bioconductor
## digest 0.6.27 2020-10-24 [1] CRAN (R 4.1.0)
## DO.db 2.9 2021-06-02 [1] Bioconductor
## DOSE 3.18.0 2021-05-19 [1] Bioconductor
## dplyr * 1.0.6 2021-05-05 [1] CRAN (R 4.1.0)
## ellipsis 0.3.2 2021-04-29 [1] CRAN (R 4.1.0)
## enrichplot 1.12.0 2021-05-19 [1] Bioconductor
## evaluate 0.14 2019-05-28 [1] CRAN (R 4.1.0)
## fansi 0.5.0 2021-05-25 [1] CRAN (R 4.1.0)
## farver 2.1.0 2021-02-28 [1] CRAN (R 4.1.0)
## fastmap 1.1.0 2021-01-25 [1] CRAN (R 4.1.0)
## fastmatch 1.1-0 2017-01-28 [1] CRAN (R 4.1.0)
## fgsea 1.18.0 2021-05-19 [1] Bioconductor
## filelock 1.0.2 2018-10-05 [1] CRAN (R 4.1.0)
## FindIT2 * 0.99.13 2021-08-11 [1] Bioconductor
## foreach 1.5.1 2020-10-15 [1] CRAN (R 4.1.0)
## generics 0.1.0 2020-10-31 [1] CRAN (R 4.1.0)
## GenomeInfoDb * 1.28.0 2021-05-19 [1] Bioconductor
## GenomeInfoDbData 1.2.6 2021-06-02 [1] Bioconductor
## GenomicAlignments 1.28.0 2021-05-19 [1] Bioconductor
## GenomicFeatures * 1.44.0 2021-05-19 [1] Bioconductor
## GenomicRanges * 1.44.0 2021-05-19 [1] Bioconductor
## ggforce 0.3.3 2021-03-05 [1] CRAN (R 4.1.0)
## ggplot2 3.3.5 2021-06-25 [1] CRAN (R 4.1.0)
## ggraph 2.0.5 2021-02-23 [1] CRAN (R 4.1.0)
## ggrepel 0.9.1 2021-01-15 [1] CRAN (R 4.1.0)
## ggtree 3.0.2 2021-06-01 [1] Bioconductor
## glmnet 4.1-1 2021-02-21 [1] CRAN (R 4.1.0)
## glue 1.4.2 2020-08-27 [1] CRAN (R 4.1.0)
## GO.db 3.13.0 2021-06-02 [1] Bioconductor
## GOSemSim 2.18.0 2021-05-19 [1] Bioconductor
## gplots 3.1.1 2020-11-28 [1] CRAN (R 4.1.0)
## graphlayouts 0.7.1 2020-10-26 [1] CRAN (R 4.1.0)
## gridExtra 2.3 2017-09-09 [1] CRAN (R 4.1.0)
## gtable 0.3.0 2019-03-25 [1] CRAN (R 4.1.0)
## gtools 3.8.2 2020-03-31 [1] CRAN (R 4.1.0)
## highr 0.9 2021-04-16 [1] CRAN (R 4.1.0)
## hms 1.1.0 2021-05-17 [1] CRAN (R 4.1.0)
## htmltools 0.5.1.1 2021-01-22 [1] CRAN (R 4.1.0)
## httr 1.4.2 2020-07-20 [1] CRAN (R 4.1.0)
## igraph 1.2.6 2020-10-06 [1] CRAN (R 4.1.0)
## IRanges * 2.26.0 2021-05-19 [1] Bioconductor
## iterators 1.0.13 2020-10-15 [1] CRAN (R 4.1.0)
## jquerylib 0.1.4 2021-04-26 [1] CRAN (R 4.1.0)
## jsonlite 1.7.2 2020-12-09 [1] CRAN (R 4.1.0)
## KEGGREST 1.32.0 2021-05-19 [1] Bioconductor
## KernSmooth 2.23-20 2021-05-03 [1] CRAN (R 4.1.0)
## knitr 1.33 2021-04-24 [1] CRAN (R 4.1.0)
## labeling 0.4.2 2020-10-20 [1] CRAN (R 4.1.0)
## lattice 0.20-44 2021-05-02 [1] CRAN (R 4.1.0)
## lazyeval 0.2.2 2019-03-15 [1] CRAN (R 4.1.0)
## lifecycle 1.0.0 2021-02-15 [1] CRAN (R 4.1.0)
## magrittr 2.0.1 2020-11-17 [1] CRAN (R 4.1.0)
## MASS 7.3-54 2021-05-03 [1] CRAN (R 4.1.0)
## Matrix 1.3-3 2021-05-04 [1] CRAN (R 4.1.0)
## MatrixGenerics 1.4.2 2021-08-08 [1] Bioconductor
## matrixStats 0.60.0 2021-07-26 [1] CRAN (R 4.1.0)
## memoise 2.0.0 2021-01-26 [1] CRAN (R 4.1.0)
## munsell 0.5.0 2018-06-12 [1] CRAN (R 4.1.0)
## nlme 3.1-152 2021-02-04 [1] CRAN (R 4.1.0)
## patchwork 1.1.1 2020-12-17 [1] CRAN (R 4.1.0)
## pillar 1.6.2 2021-07-29 [1] CRAN (R 4.1.0)
## pkgconfig 2.0.3 2019-09-22 [1] CRAN (R 4.1.0)
## plotrix 3.8-1 2021-01-21 [1] CRAN (R 4.1.0)
## plyr 1.8.6 2020-03-03 [1] CRAN (R 4.1.0)
## png 0.1-7 2013-12-03 [1] CRAN (R 4.1.0)
## polyclip 1.10-0 2019-03-14 [1] CRAN (R 4.1.0)
## prettyunits 1.1.1 2020-01-24 [1] CRAN (R 4.1.0)
## progress 1.2.2 2019-05-16 [1] CRAN (R 4.1.0)
## purrr 0.3.4 2020-04-17 [1] CRAN (R 4.1.0)
## qvalue 2.24.0 2021-05-19 [1] Bioconductor
## R6 2.5.0 2020-10-28 [1] CRAN (R 4.1.0)
## rappdirs 0.3.3 2021-01-31 [1] CRAN (R 4.1.0)
## RColorBrewer 1.1-2 2014-12-07 [1] CRAN (R 4.1.0)
## Rcpp 1.0.6 2021-01-15 [1] CRAN (R 4.1.0)
## RCurl 1.98-1.3 2021-03-16 [1] CRAN (R 4.1.0)
## reshape2 1.4.4 2020-04-09 [1] CRAN (R 4.1.0)
## restfulr 0.0.13 2017-08-06 [1] CRAN (R 4.1.0)
## rjson 0.2.20 2018-06-08 [1] CRAN (R 4.1.0)
## rlang 0.4.11 2021-04-30 [1] CRAN (R 4.1.0)
## rmarkdown 2.8 2021-05-07 [1] CRAN (R 4.1.0)
## Rsamtools 2.8.0 2021-05-19 [1] Bioconductor
## RSQLite 2.2.7 2021-04-22 [1] CRAN (R 4.1.0)
## rstudioapi 0.13 2020-11-12 [1] CRAN (R 4.1.0)
## rtracklayer 1.52.0 2021-05-19 [1] Bioconductor
## rvcheck 0.1.8 2020-03-01 [1] CRAN (R 4.1.0)
## S4Vectors * 0.30.0 2021-05-19 [1] Bioconductor
## sass 0.4.0 2021-05-12 [1] CRAN (R 4.1.0)
## scales 1.1.1 2020-05-11 [1] CRAN (R 4.1.0)
## scatterpie 0.1.6 2021-04-23 [1] CRAN (R 4.1.0)
## sessioninfo 1.1.1 2018-11-05 [1] CRAN (R 4.1.0)
## shadowtext 0.0.8 2021-04-23 [1] CRAN (R 4.1.0)
## shape 1.4.6 2021-05-19 [1] CRAN (R 4.1.0)
## stringi 1.6.2 2021-05-17 [1] CRAN (R 4.1.0)
## stringr 1.4.0 2019-02-10 [1] CRAN (R 4.1.0)
## SummarizedExperiment 1.22.0 2021-05-19 [1] Bioconductor
## survival 3.2-11 2021-04-26 [1] CRAN (R 4.1.0)
## tibble 3.1.2 2021-05-16 [1] CRAN (R 4.1.0)
## tidygraph 1.2.0 2020-05-12 [1] CRAN (R 4.1.0)
## tidyr 1.1.3 2021-03-03 [1] CRAN (R 4.1.0)
## tidyselect 1.1.1 2021-04-30 [1] CRAN (R 4.1.0)
## tidytree 0.3.4 2021-05-22 [1] CRAN (R 4.1.0)
## treeio 1.16.1 2021-05-23 [1] Bioconductor
## tweenr 1.0.2 2021-03-23 [1] CRAN (R 4.1.0)
## TxDb.Athaliana.BioMart.plantsmart28 * 3.2.2 2021-06-07 [1] Bioconductor
## TxDb.Hsapiens.UCSC.hg19.knownGene 3.2.2 2021-06-02 [1] Bioconductor
## utf8 1.2.1 2021-03-12 [1] CRAN (R 4.1.0)
## vctrs 0.3.8 2021-04-29 [1] CRAN (R 4.1.0)
## viridis 0.6.1 2021-05-11 [1] CRAN (R 4.1.0)
## viridisLite 0.4.0 2021-04-13 [1] CRAN (R 4.1.0)
## withr 2.4.2 2021-04-18 [1] CRAN (R 4.1.0)
## xfun 0.23 2021-05-15 [1] CRAN (R 4.1.0)
## XML 3.99-0.6 2021-03-16 [1] CRAN (R 4.1.0)
## xml2 1.3.2 2020-04-23 [1] CRAN (R 4.1.0)
## XVector 0.32.0 2021-05-19 [1] Bioconductor
## yaml 2.2.1 2020-02-01 [1] CRAN (R 4.1.0)
## zlibbioc 1.38.0 2021-05-19 [1] Bioconductor
##
## [1] E:/R/R-4.1.0/libraryReference
Wang, F.-X., Shang, G.-D., Wu, L.-Y., Xu, Z.-G., Zhao, X.-Y., and Wang, J.-W. (2020). Chromatin Accessibility Dynamics and a Hierarchical Transcriptional Regulatory Network Structure for Plant Somatic Embryogenesis. Developmental Cell 54, 742-757.e8.↩︎