Intro_FindIT2:find_influential_TF module part3
前两篇文章介绍了如何寻找调控你感兴趣feature的那些TF,下面两篇文章将会来介绍如何对那些TF进行趋势变化的探究。这篇文章首先来讲如何使用findIT_MARA函数对感兴趣的peak做TF的趋势变化研究。
Pre-preparation
这次的测试数据没有那么复杂,全部是围绕多时间的ATAC-seq相关的数据。
# 加载包
library(FindIT2)
library(TxDb.Athaliana.BioMart.plantsmart28)
library(BSgenome.Athaliana.TAIR.TAIR9)
library(motifmatchr)
library(dplyr)
library(ggplot2)
library(ComplexHeatmap)
library(circlize)
Txdb <- TxDb.Athaliana.BioMart.plantsmart28
seqlevels(Txdb) <- paste0("Chr", c(1:5, "M", "C"))我们重新下载一遍需要用到的数据
# 下载ATAC的merge peak文件
download.file(url = "https://raw.githubusercontent.com/shangguandong1996/FindIT2_paper_relatedCode/master/rawdata/ATAC/ATAC_mergePeak.bed",
destfile = "ATAC_mergePeak.bed")
# 下载ATAC merge peak文件对应的多时间count矩阵
# 这里的count矩阵已经经过了DESeq2的counts函数的矫正
download.file(url = "https://raw.githubusercontent.com/shangguandong1996/FindIT2_paper_relatedCode/master/rawdata/ATAC/ATAC_peakNormCount.rda",
destfile = "ATAC_peakNormCount.rda"
)
# 下载ATAC peak的DESeq2 LRT分析结果
# 这里的分析结果主要是为了筛选出变化最大的Top peak
download.file(url = "https://raw.githubusercontent.com/shangguandong1996/FindIT2_paper_relatedCode/master/rawdata/ATAC/ATAC_LRT_result.rda",
destfile = "ATAC_LRT_result.rda")展示下载数据
ATAC_peak_GR <- loadPeakFile("ATAC_mergePeak.bed")
ATAC_peak_GR
## GRanges object with 22994 ranges and 2 metadata columns:
## seqnames ranges strand | feature_id score
## <Rle> <IRanges> <Rle> | <character> <numeric>
## [1] Chr1 30-875 * | ATAC_1 0
## [2] Chr1 1392-1953 * | ATAC_2 0
## [3] Chr1 2164-3625 * | ATAC_3 0
## [4] Chr1 8268-8966 * | ATAC_4 0
## [5] Chr1 13654-14698 * | ATAC_5 0
## ... ... ... ... . ... ...
## [22990] Chr5 26969184-26969915 * | ATAC_22990 0
## [22991] Chr5 26970542-26971504 * | ATAC_22991 0
## [22992] Chr5 26972304-26973186 * | ATAC_22992 0
## [22993] Chr5 26974280-26974825 * | ATAC_22993 0
## [22994] Chr5 26975157-26975438 * | ATAC_22994 0
## -------
## seqinfo: 5 sequences from an unspecified genome; no seqlengthsload("ATAC_peakNormCount.rda")
head(peakCountNorm_ATAC)
## WT_E5_0h_R1 WT_E5_0h_R2 WT_E5_4h_R1 WT_E5_4h_R2 WT_E5_8h_R1 WT_E5_8h_R2
## ATAC_1 126.65266 143.0386 220.6055 224.7059 171.1754 204.2058
## ATAC_2 93.45245 103.4747 106.0053 132.7808 151.9182 152.2089
## ATAC_3 354.75043 402.9427 621.7065 569.9358 487.8500 406.5208
## ATAC_4 158.62324 144.2559 183.3605 183.8503 165.8262 180.5708
## ATAC_5 222.56439 258.6868 203.4155 275.7754 171.1754 192.8610
## ATAC_6 201.04574 175.9070 137.5203 134.8235 145.4991 119.1200
## WT_E5_16h_R1 WT_E5_16h_R2 WT_E5_24h_R1 WT_E5_24h_R2 WT_E5_48h_R1
## ATAC_1 184.5536 152.9906 205.0690 182.4882 126.4342
## ATAC_2 153.0956 178.4891 204.3619 184.2152 105.5451
## ATAC_3 440.4120 413.7701 506.3083 472.0515 307.8398
## ATAC_4 191.8938 119.3790 221.3331 190.5476 180.3062
## ATAC_5 197.1368 200.5104 208.6047 223.3609 133.0308
## ATAC_6 139.4638 114.7430 185.2692 156.0073 124.2354
## WT_E5_48h_R2 WT_E5_48h_R3 WT_E5_72h_R1 WT_E5_72h_R2 WT_E5_72h_R3
## ATAC_1 128.4671 151.36817 134.9386 158.33027 112.9270
## ATAC_2 117.0983 97.89572 84.6225 119.51381 134.0553
## ATAC_3 345.6106 403.10002 408.2464 362.62738 446.6083
## ATAC_4 120.5090 176.04777 137.2257 150.15838 143.5266
## ATAC_5 172.8053 166.99858 152.0918 196.12523 214.9257
## ATAC_6 120.5090 106.12225 101.7757 79.67588 129.6840# 这里padj越小,说明这个peak在多个时间点变化越大
load("ATAC_LRT_result.rda")
head(LRT_result)
## # A tibble: 6 x 2
## feature_id padj
## <chr> <dbl>
## 1 ATAC_1 0.0000921
## 2 ATAC_2 0.000000436
## 3 ATAC_3 0.00000339
## 4 ATAC_4 0.144
## 5 ATAC_5 0.00533
## 6 ATAC_6 0.000143findIT_MARA
findIT_MARA基于的是2009年一篇文章1中提出的Motif Activity Response Analysis(MARA)模型,基本原理就是用peak count作为因变量,TF motif作为自变量,然后想用TF motif去解释peak count值。这里本质上是一个多元线性回归模型。这里的回归拟合基于的是ridge regression,即岭回归。之所以选择岭回归而非LASSO回归是因为我目的是去解释模型,而非筛选变量。这里我认为每个TF motif,即每个变量都会对peak count有贡献,只不过有些TF影响大,有些TF影响小。当然,也是因为findIT_MARA所借鉴的另一个软件GimmeMotifs里面的maelstrom2也是用的岭回归。
在得到了每个TF对应的参数值之后,参数值会经过排序,变换,然后最终产生TF在这个样本里面的TF activity。这里参数值的大小就跟普通的回归一样,如果某一个TF在count低的peak中几乎没有motif,而在count值高的motif有很多,那么说明该TF跟这些peak count呈现正相关,参数值就会很高,最终的TF activity也会很高。反之,就会呈现负相关,参数值就会很低,TF activity也会很低。
findIT_MARA需要的是你的目的peak集合、全部的ATAC peak GRange、全部的peak对应的多时间count矩阵以及在你的全部peak上的TF motif scan结果。
前面我们已经下载了全部的ATAC peak位置,peak对应的多时间点count矩阵。所以下面需要确定的是目的peak集合以及TF motif scan结果。
这里我还是用motifmatchr来得到ATAC peak上的motif scan结果。当然,你也可以用GimmeMotifs、HOMER、MEME、MOODS等软件来得到ATAC peak上的motif scan结果,然后产生的bed文件再导入成GRanges格式。有一点需要注意的是对于这个函数,我们不能像前面找感兴趣TF那样使用公共数据库,因为我这里本质上是用TF motif去拟合回归模型,每个TF之间都是有关联的。而公共数据库每个TF_id对应的结果是没有关联的。
getJasparMotifs <- function(species = "Arabidopsis thaliana",
collection = "CORE", ...) {
opts <- list()
opts["species"] <- species
opts["collection"] <- collection
opts <- c(opts, list(...))
out <- TFBSTools::getMatrixSet(JASPAR2020::JASPAR2020, opts)
if (!isTRUE(all.equal(TFBSTools::name(out), names(out))))
names(out) <- paste(names(out), TFBSTools::name(out), sep = "_")
return(out)
}
motifs_JASPAR2020 <- getJasparMotifs()
# call motif
motif_ix <- matchMotifs(motifs_JASPAR2020,
ATAC_peak_GR,
genome = BSgenome.Athaliana.TAIR.TAIR9,
out = "positions",
bg = "genome")
motif_GR <- unlist(motif_ix)# 再次注意要添加一列TF_id标识你的TF信息
motif_GR$TF_id <- gsub("(MA[0-9]{4}\\.[0-9]_)", "", names(motif_GR))
motif_GR
## GRanges object with 2147996 ranges and 2 metadata columns:
## seqnames ranges strand | score TF_id
## <Rle> <IRanges> <Rle> | <numeric> <character>
## MA0121.1_ARR10 Chr1 1490-1497 - | 10.9848 ARR10
## MA0121.1_ARR10 Chr1 1904-1911 - | 10.9848 ARR10
## MA0121.1_ARR10 Chr1 33532-33539 - | 10.9848 ARR10
## MA0121.1_ARR10 Chr1 55227-55234 - | 11.1123 ARR10
## MA0121.1_ARR10 Chr1 62176-62183 - | 11.1123 ARR10
## ... ... ... ... . ... ...
## MA1329.2_ZHD1 Chr5 26876218-26876230 - | 18.9609 ZHD1
## MA1329.2_ZHD1 Chr5 26879928-26879940 - | 19.6284 ZHD1
## MA1329.2_ZHD1 Chr5 26880196-26880208 - | 19.6284 ZHD1
## MA1329.2_ZHD1 Chr5 26889355-26889367 - | 20.1565 ZHD1
## MA1329.2_ZHD1 Chr5 26901566-26901578 - | 19.1066 ZHD1
## -------
## seqinfo: 5 sequences from an unspecified genome; no seqlengths# 跟前面的integrate_ChIP_RNA函数类似
# 如果你的GRanges对象里面有feature_score这一列
# 那么最终的findIT_MARA就会将这一列考虑在内
# 这里的feature_score指的是TF motif scan的置信度
# 如果score越高,越说明这几个bp越有可能是这个TF的motif
motif_GR$feature_score <- motif_GR$score对于目的peak的集合,我选择Top15000变化最大的peak。理论上来说通过这些peak所产生的TF activity就可以表现全局性的TF变化了。
LRT_result %>%
arrange(padj) %>%
slice(1:15000) %>%
pull(feature_id) -> input_feature_id然后就可以开始分析了。
# 因为我是用glmnet包的glmnet函数去做岭回归
# 这里面glmnet函数的lambda我是靠函数自己拟合出来的
# 所以要加种子数来保证结果一致性
set.seed(20160806)
findIT_MARA(input_feature_id = input_feature_id,
peak_GR = ATAC_peak_GR,
peakScoreMt = peakCountNorm_ATAC,
TF_GR_database = motif_GR) -> result_MARA
## >> dealing with TF_GR_database... 2021-11-02 10:00:30
## >> calculating coef and converting into z-score using INT... 2021-11-02 10:01:06
## >> dealing withWT_E5_0h_R1... 2021-11-02 10:01:06
## >> dealing withWT_E5_0h_R2... 2021-11-02 10:01:29
## >> dealing withWT_E5_4h_R1... 2021-11-02 10:01:50
## >> dealing withWT_E5_4h_R2... 2021-11-02 10:02:12
## >> dealing withWT_E5_8h_R1... 2021-11-02 10:02:33
## >> dealing withWT_E5_8h_R2... 2021-11-02 10:02:54
## >> dealing withWT_E5_16h_R1... 2021-11-02 10:03:15
## >> dealing withWT_E5_16h_R2... 2021-11-02 10:03:36
## >> dealing withWT_E5_24h_R1... 2021-11-02 10:03:57
## >> dealing withWT_E5_24h_R2... 2021-11-02 10:04:19
## >> dealing withWT_E5_48h_R1... 2021-11-02 10:04:39
## >> dealing withWT_E5_48h_R2... 2021-11-02 10:05:00
## >> dealing withWT_E5_48h_R3... 2021-11-02 10:05:22
## >> dealing withWT_E5_72h_R1... 2021-11-02 10:05:42
## >> dealing withWT_E5_72h_R2... 2021-11-02 10:06:04
## >> dealing withWT_E5_72h_R3... 2021-11-02 10:06:25
## >> merging all info together... 2021-11-02 10:06:46
## >> done 2021-11-02 10:06:46
result_MARA
## # A tibble: 465 x 17
## TF_id WT_E5_0h_R1 WT_E5_0h_R2 WT_E5_4h_R1 WT_E5_4h_R2 WT_E5_8h_R1 WT_E5_8h_R2
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ARR10 -1.39 -1.26 0.135 -0.108 -0.845 -0.206
## 2 BZR2 0.206 -0.639 0.463 0.476 0.537 0.234
## 3 HY5 0.562 0.549 0.387 0.494 0.273 0.119
## 4 PIF1 -0.543 -0.556 -0.152 -0.234 0.262 0.301
## 5 SMZ 0.861 -1.27 -1.66 -1.40 -1.64 -1.81
## 6 SOC1 0.885 0.771 0.512 0.771 0.0701 0.500
## 7 SVP -0.103 -0.476 0.537 0.251 -0.341 0.245
## 8 AP3 -0.184 0.295 0.815 0.707 -0.352 0.0755
## 9 FHY3 0.434 -0.686 -0.756 -1.32 0.680 -0.934
## 10 FLC -0.146 0.500 0.877 0.950 -0.793 -0.463
## # ... with 455 more rows, and 10 more variables: WT_E5_16h_R1 <dbl>,
## # WT_E5_16h_R2 <dbl>, WT_E5_24h_R1 <dbl>, WT_E5_24h_R2 <dbl>,
## # WT_E5_48h_R1 <dbl>, WT_E5_48h_R2 <dbl>, WT_E5_48h_R3 <dbl>,
## # WT_E5_72h_R1 <dbl>, WT_E5_72h_R2 <dbl>, WT_E5_72h_R3 <dbl>result_MARA中存放的就是每个TF在各个时间点的TF activity的变化。我们可以挑选变化最大的前40个TF,然后用热图进行展示。
MARA_mt <- as.matrix(result_MARA[, -1])
rownames(MARA_mt) <- result_MARA$TF_id
apply(MARA_mt, 1, sd) %>%
sort(decreasing = TRUE) %>%
names() %>%
"["(1:40) -> TF_name
col_fun_matrix <- colorRamp2(seq(-3, 3, length.out = 9),
as.character(BuenColors::jdb_palette("solar_extra")))
Heatmap(MARA_mt[TF_name, ],
col = col_fun_matrix,
cluster_rows = TRUE,
cluster_columns = FALSE,
rect_gp = gpar(col = "white", lwd = 2),
border = TRUE,
) -> hmhm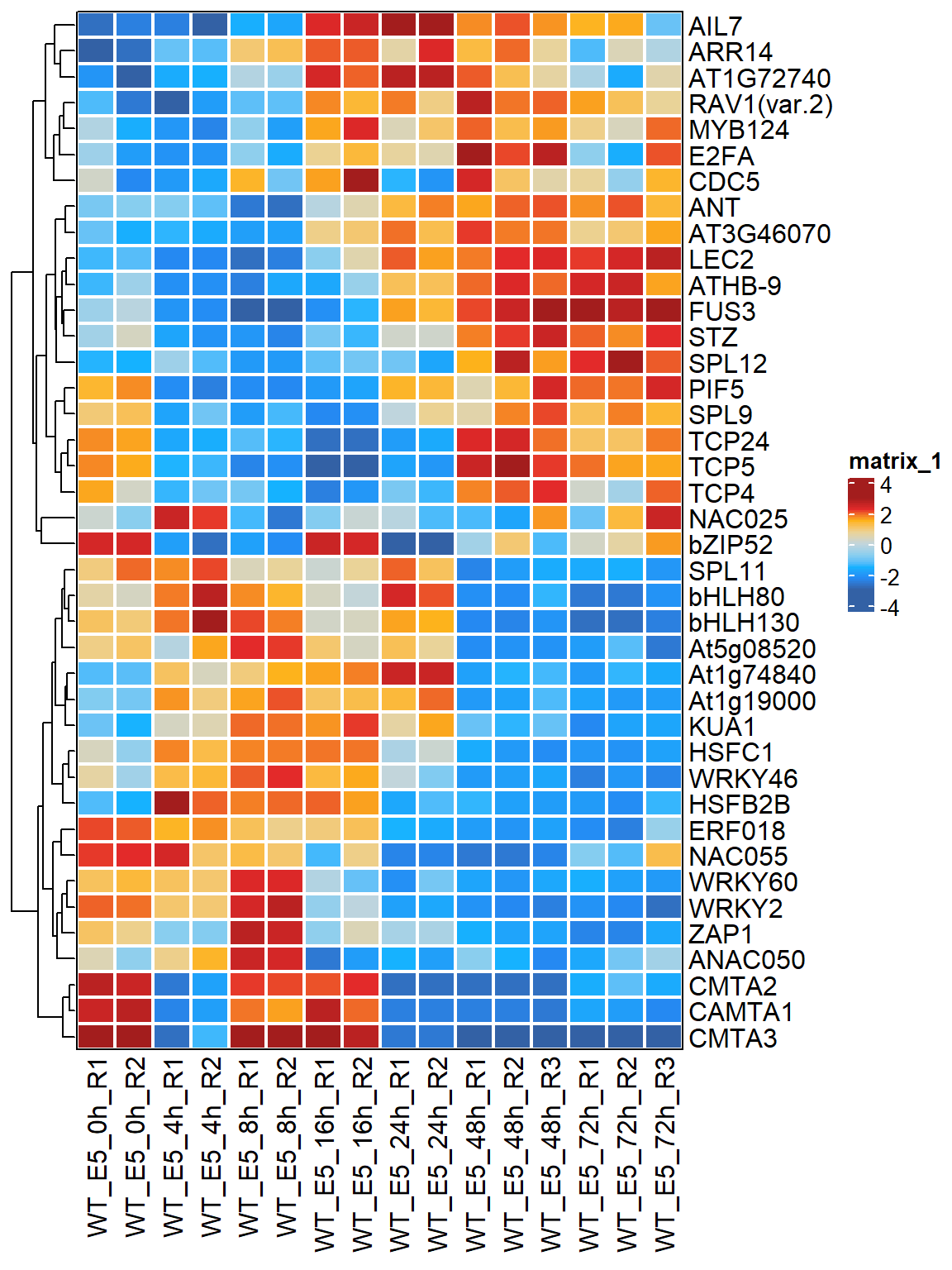
考虑到这里每个时间点有2-3个重复,最后的结果展示有点过于冗长。所以我们可以用integrate_replicates函数来整合重复内的TF activity。不过在整合前还是建议先看看原始结果,有些TF的结果可能在重复内的差异有些大。
integrate_replicates可以整合不同类型的数值,包括像数值、rank、p-value等。这部分会在FindIT2的“番外篇”提到
# integrate_replicates需要三个参数值
# 第一个参数值是你的样本信息,即一个叫做coldata的数据框
# 该数据框只有一列(仿照的是DESeq2的coldata)
# 列名必须是叫type,type里面是每个重复对应的样本类型
# 行名是你要整合重复的矩阵的列名
colData <- data.frame(row.names = colnames(MARA_mt),
type = gsub("_R[0-9]", "",
colnames(MARA_mt)))
colData
## type
## WT_E5_0h_R1 WT_E5_0h
## WT_E5_0h_R2 WT_E5_0h
## WT_E5_4h_R1 WT_E5_4h
## WT_E5_4h_R2 WT_E5_4h
## WT_E5_8h_R1 WT_E5_8h
## WT_E5_8h_R2 WT_E5_8h
## WT_E5_16h_R1 WT_E5_16h
## WT_E5_16h_R2 WT_E5_16h
## WT_E5_24h_R1 WT_E5_24h
## WT_E5_24h_R2 WT_E5_24h
## WT_E5_48h_R1 WT_E5_48h
## WT_E5_48h_R2 WT_E5_48h
## WT_E5_48h_R3 WT_E5_48h
## WT_E5_72h_R1 WT_E5_72h
## WT_E5_72h_R2 WT_E5_72h
## WT_E5_72h_R3 WT_E5_72h
# 第二个参数值是待处理的矩阵
# 注意这里的mt是通过as.matrix(result_MARA[, -1])得到的矩阵
# 而不是最开始findIT_MARA得到的结果
head(MARA_mt)
## WT_E5_0h_R1 WT_E5_0h_R2 WT_E5_4h_R1 WT_E5_4h_R2 WT_E5_8h_R1 WT_E5_8h_R2
## ARR10 -1.3865094 -1.2574185 0.1351755 -0.1080217 -0.84546823 -0.2062967
## BZR2 0.2062967 -0.6393725 0.4634934 0.4755309 0.53681147 0.2339114
## HY5 0.5618862 0.5493057 0.3867724 0.4937180 0.27287899 0.1188725
## PIF1 -0.5430480 -0.5555849 -0.1515145 -0.2339114 0.26170565 0.3009654
## SMZ 0.8609832 -1.2693926 -1.6606976 -1.4007451 -1.63966294 -1.8056838
## SOC1 0.8846525 0.7707414 0.5120698 0.7707414 0.07013524 0.4998165
## WT_E5_16h_R1 WT_E5_16h_R2 WT_E5_24h_R1 WT_E5_24h_R2 WT_E5_48h_R1
## ARR10 -0.1080217 -0.2228453 0.78533349 1.3192164 1.1569034
## BZR2 0.3694022 0.6932191 -0.15151448 0.1026011 -0.3009654
## HY5 -0.6526553 -0.6262015 -0.79269252 -0.7069945 0.2007933
## PIF1 0.1460639 0.3925882 0.56820987 0.3349876 0.3292913
## SMZ 1.9645843 1.5996447 -0.05393212 0.3809696 2.2626720
## SOC1 -0.6066458 -0.5937368 -0.88465249 -0.3694022 -0.5809260
## WT_E5_48h_R2 WT_E5_48h_R3 WT_E5_72h_R1 WT_E5_72h_R2 WT_E5_72h_R3
## ARR10 0.6262015 0.53059577 1.1057905 1.01139386 -0.51822531
## BZR2 -0.1843187 -0.13517550 -0.9847820 -0.39841734 -0.35214279
## HY5 0.1843187 -0.01078141 -0.2339114 -0.41011658 0.21180641
## PIF1 0.5059337 0.21732254 -0.3694022 -0.06473236 0.09176882
## SMZ 1.0295404 1.75318712 -0.9674216 -1.26939260 2.19788939
## SOC1 -0.6196566 0.34641313 -0.5682099 -0.52440051 0.08635679
# 第三个参数值是你待处理矩阵的type类型
# 这里的数值类型是inverse normal transformation(INT)变换后的值
# 所以type选择"rank_zscore"
integrate_replicates(mt = MARA_mt,
colData = colData,
type = "rank_zscore") -> MARA_mt_merge再一次地,我们可以用热图来展现结果
Heatmap(MARA_mt_merge[TF_name, ],
col = col_fun_matrix,
cluster_rows = TRUE,
cluster_columns = FALSE,
rect_gp = gpar(col = "white", lwd = 2),
border = TRUE,
) -> hm# 这样结果就整洁多了
hm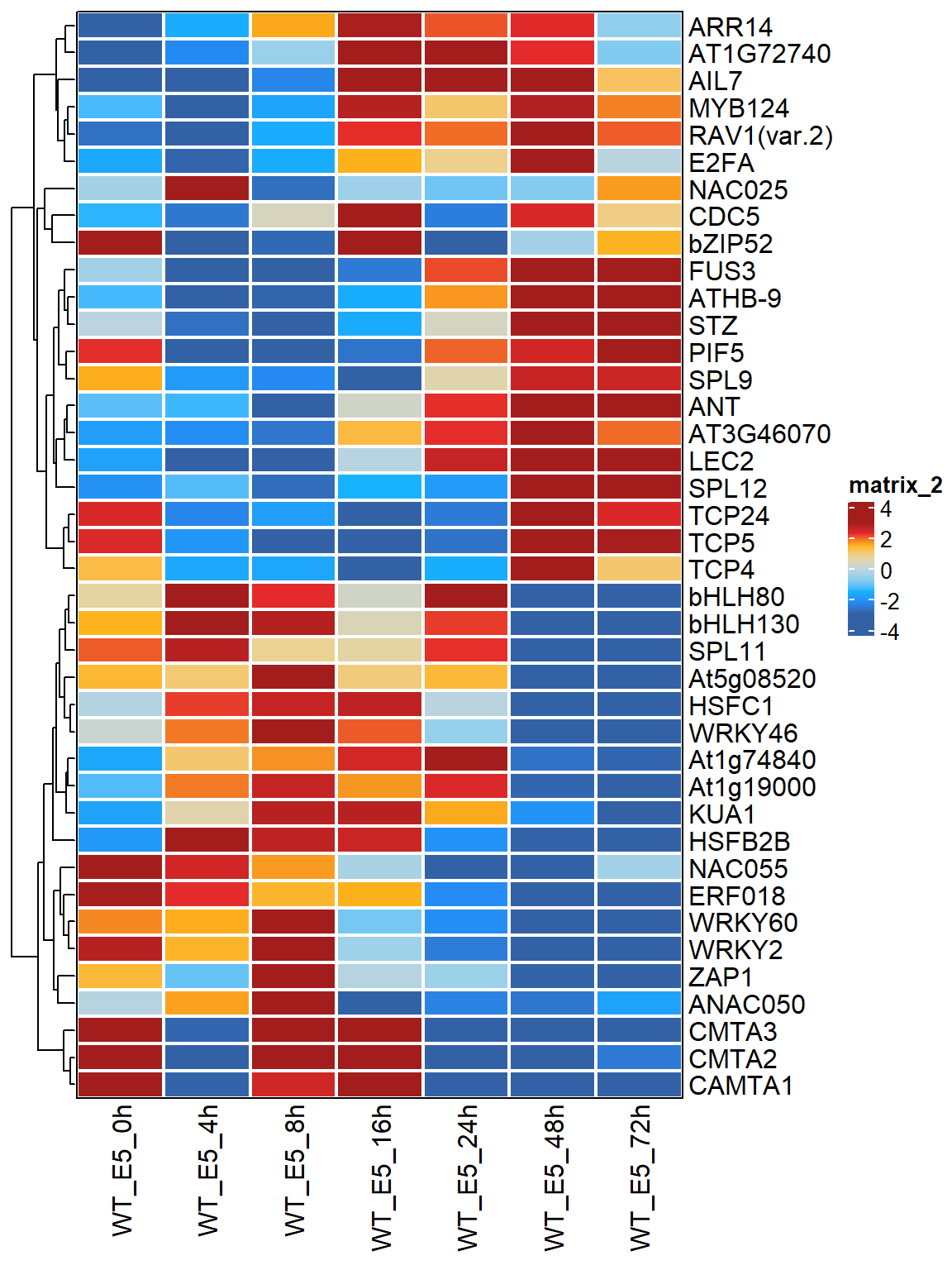
可以看到LEC2的TF activity主要在后期比较高。
同样地,还有一些注意点需要考虑。首先就是由于同一家族内的TF可能存在着motif的相似性,举例而言同属于B3家族的FUS3和ABI3就非常的相似,所以在得到了TF的activity或者说前面得到的TF富集之后,还需要同时参考TF的表达量来进一步地锁定真正发挥功能的TF。
其次就是你会发现某些TF的activity在所有时间点都不是很高,这可能并不是这个TF没有功能,而是说这个TF在大部分peak中都是富集的。在count高的peak中这些TF也有很多motif hit,在count低的peak中也有很多motif hit,那么自然就导致TF跟peak count高低的相关性并不是很高。但我们并不能说这些TF不重要。
最后就是这篇文章虽然讲的是对感兴趣的peak做TF的趋势变化研究,但无论是所借鉴的MARA模型,还是所借鉴的软件gimmemotifs,其所产生的TF activity针对的都是全局性的,即感兴趣的peak是全部的peak或者说变化最剧烈的top peak。所以可以看到我例子里面也是用的top 15000 peak。从我个人角度来看,findIT_MARA单纯应用于感兴趣的peak集合,就像上一篇文章用到的LEC2相关的peak集合似乎是没有问题的,但具体的实践我还没有试过,这也是我个人的局限性所在╮(╯_╰)╭。所以总体来说,如果你想用这个函数看下全局性的TF activity变化,是没有问题的,但如果只想要看下一部分peak中TF activity,可能需要自己测试下,也欢迎大家提出自己的意见。
Reference
FANTOM Consortium and Riken Omics Science Center (2009). The transcriptional network that controls growth arrest and differentiation in a human myeloid leukemia cell line. Nat Genet 41, 553–562.↩︎